9月10日,我校附属第二医院病毒性肝炎研究所沈伟副研究员以第一作者和共同通讯作者身份与欧洲生物信息研究所(EMBL-EBI)Zamin Iqbal教授合作,在国际权威期刊Nature Biotechnology在线发表题为Efficient sequence alignment against millions of prokaryotic genomes with LexicMap的原创性研究论文。

如同使用搜索引擎在互联网上查找网页一样,生物学家也常常需要在已有测序数据中搜索感兴趣的生物序列。然而,由于进化和突变的存在,查询序列和目标序列往往存在差异,从而需要复杂的计算——这个过程称为“序列比对”(Sequence Alignment)。近年来,全球微生物序列数据持续、快速增长,其规模远超现有序列比对软件的计算能力。
本研究中,作者开发了一个全新的序列比对软件LexicMap,支持在百万规模原核(细菌与古菌)基因组中,对基因、质粒、长读长测序数据进行准确、快速、低内存的碱基水平序列比对。与现有方法(包括Blastn、Minimap2、MMseqs2、Ropebwt3)相比,LexicMap在保持相当准确性的同时,速度更快、内存占用更低、具有更高的可扩展性。
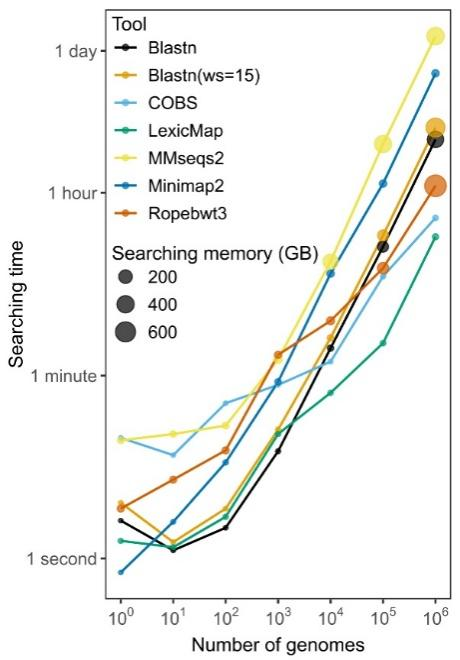
序列比对/搜索性能比较
该研究受国家自然科学基金、国家留学基金委、EMBL Visitor/Sabbatical Programme fellowship、重医附二院登峰学科群联合项目和宽仁英才等项目资助。全文链接:https://www.nature.com/articles/s41587-025-02812-8
作者介绍

沈伟,理学博士,副研究员,硕士生导师(生物信息学),重庆生物信息学会常务理事。主要研究方向为微生物基因组、宏基因组、大规模基因组数据检索等生物信息学算法设计与软件开发。主持国家自然科学基金面上项目等科研项目5项;以第一和/或通讯作者身份在Nature Biotechnology、iMeta、Bioinformatics等期刊发表SCI论文9篇,其中2篇为ESI高被引论文,单篇最高引用2800余次。沈伟课题组网站:https://mbio.info/cn/